EL PROBLEMA
La Resonancia Magnética Funcional (o fMRI) es una técnica de neuroimagen no-invasiva que puede ser usada para estudiar la funcionalidad del cerebro. La modalidad de señal de fMRI más común es la señal BOLD (dependiente del nivel de oxígeno en la sangre, o en inglés Blood-oxygen-level dependent), relacionada con cambios locales en la concentración de deoxihemoglobina en la sangre.
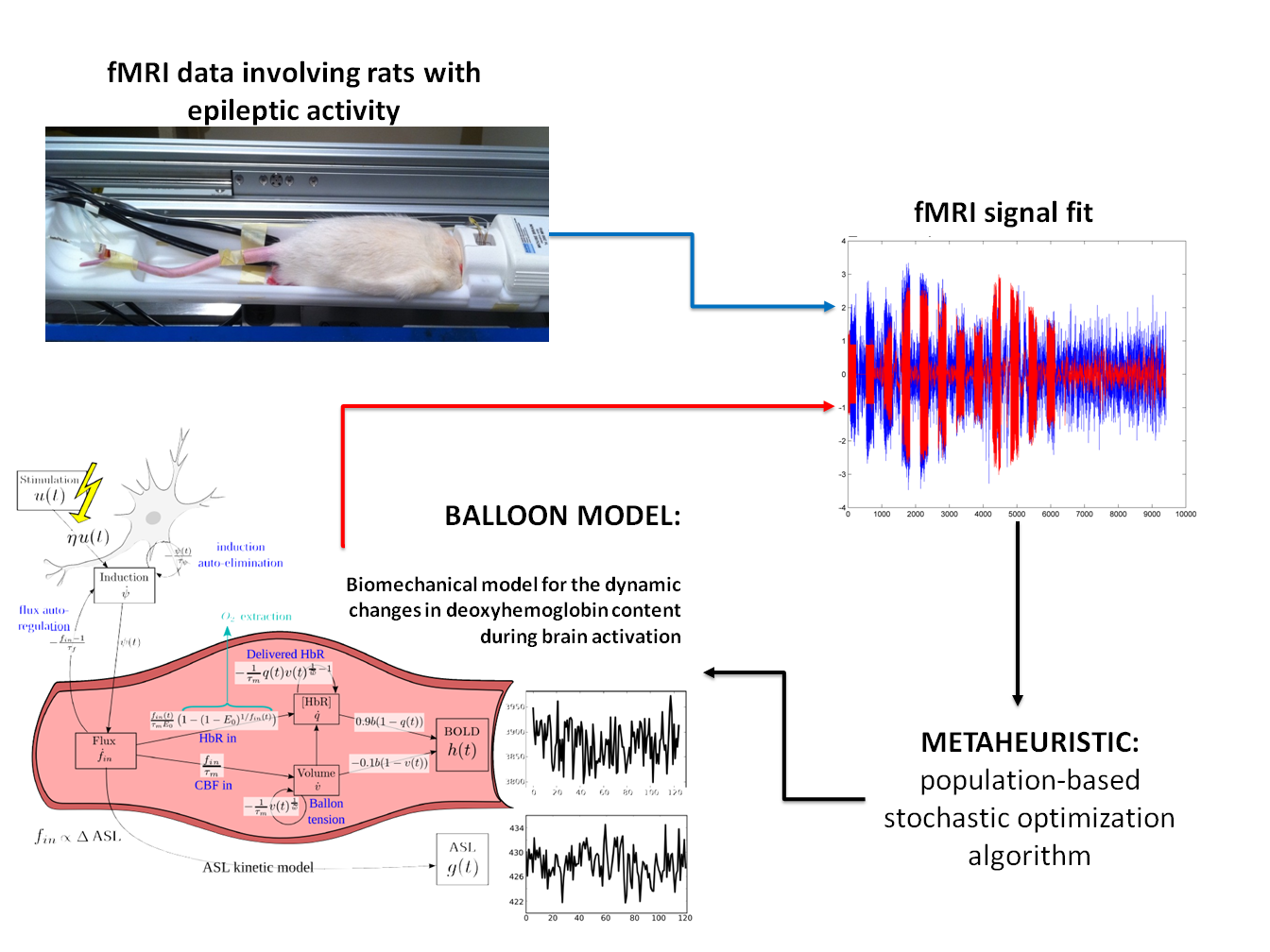
Durante los últimos 15 años, diferentes versiones del ampliamente usado «Modelo del Balón» («Balloon model») han sido introducidas para proporcionar una descripción de alto nivel de los procesos fisiológicos subyacentes a la respuesta hemodinámica, desde la activación neuronal a la señal BOLD. Dicho modelo depende de varios parámetros biofísicos (entorno a 15), para los cuales diferentes combinaciones de valores han sido propuestos en la literatura científica.
La mayor parte de los métodos existentes utilizan uno de estos conjuntos de valores empíricos, o realizan la estimación utilizando métodos basados en el gradiente a partir de una serie de valores medios escogidos a priori (y cuya elección no es trivial). En este caso concreto, las señales BOLD empleadas fueron registradas para validar modelos biofísicos en el contexto de la actividad epiléptica en ratas.
MÉTODOS EMPLEADOS
De cara a superar las limitaciones de los procedimientos tradicionalmente empleados para estimar parámetros biofísicos, empleamos un método de computación evolutiva denominado evolución diferencial (en inglés, differential evolution). Los métodos de computación evolutiva, pertenecientes a la familia de las metaheurísticas, son métodos basados en poblaciones, estocásticos, y de propósito general diseñados para resolver complejos problemas de optimización, en donde los métodos enumerativos o basados en gradiente no son apropiados. Dichos métodos evolutivos requieren la definición de una función de ajuste que mide cómo de buenos son los parámetros encontrados. Para derivar dicha función de ajuste se empleó modelado Bayesiano. Finalmente, para paliar la ausencia de ground truth (es decir, la solución correcta que uno espera encontrar al resolver el problema) en datos reales, un conjunto de datos sintéticos fue creado para estudiar el comportamiento de los distintos métodos que se compararon bajo condiciones controladas.
RESULTADOS
Se diseñó, implementó y validó (comparándolo con otros métodos existentes) un nuevo método para inferir parámetros biofísicos a partir de las señales BOLD observadas, mostrando la robustez y flexibilidad de los métodos de optimización global mientras que, a la vez, presentaba la capacidad de introducir información a priori de modo Bayesiano. El método desarrollado no necesita proporcionar valores iniciales ni la computación de derivadas. El nuevo método proporcionó un mejor ajuste de la señal en la mayor parte de los casos, y las estimaciones obtenidas resultaron más realistas y más estables entre sesiones, o al menos igualmente estables, que las proporcionadas por los métodos basados en gradiente. Los resultados obtenidos fueron publicados en una revista de referencia en procesado de señales (Mesejo, P., Saillet, S., David, O., Bénar, C., Warnking, J., and Forbes, F. (2016), A Differential Evolution-based Approach for Fitting a Nonlinear Biophysical Model to fMRI BOLD Data, IEEE Journal of Selected Topics in Signal Processing 10 (2), 416-427) y en, posiblemente, la conferencia más prestigiosa y competitiva del mundo en lo que atañe a análisis de imagen médica (MICCAI: Mesejo, P., Saillet, S., David, O., Bénar, C., Warnking, J., and Forbes, F., Estimating Biophysical Parameters from BOLD Signals through Evolutionary-based Optimization, 18th International Conference on Medical Image Computing and Computer Assisted Intervention (MICCAI’15), Part II, 528-535, Munich, 2015). El software desarrollado fue registrado en Francia el 20/07/2016 (número de referencia: IDDN.FR.001.300022.000.S.P.2016.000.31230. BOLD model FIT). El software también está disponible como MATLAB toolbox en https://hal.inria.fr/hal-01221115v2/file/fMRImodelFIT_v1.zip.
PARTICIPANTES
Instituciones
Instituto Nacional de Investigación en Informática y Automática (Institut National de Recherche en Informatique et en Automatique, Inria, Francia) e Instituto de Neurociencias de Grenoble (Grenoble Institut des Neurosciences, GIN, Francia). Trabajo financiado a través de un proyecto Inria titulado «Modelos estadísticos avanzados para el acople de modalidades de resonancia magnética ASL y BOLD para el estudio de la funcionalidad cerebral» financiado con 62.904€.
Investigadores
Dr. Pablo Mesejo como investigador postdoctoral en el equipo MISTIS (Modelado e Inferencia de Sistemas Estocásticos Complejos y Estructurados), liderado por la Dra. Florence Forbes, perteneciente al centro de investigación Inria Grenoble Rhône-Alpes (Francia). Proyecto desarrollado en colaboración con el Dr. Jan M. Warnking, investigador del Instituto de Neurociencias de Grenoble (GIN).